基本信息
期刊:Nature Communications
ABclonal产品:H3K4me1(A2355),H3K4me3(A2357),H3K27ac(A7253),H3K27me3(A2363)
物种:玉米
应用:Long-read ChIA-PET,CHIP-Seq
1、研究背景
玉米是世界上最重要的粮食作物之一,研究表明,玉米基因组大部分区域都为非编码区,且QTL和GWAS分析结果显示,玉米品种的表型差异大部分都来源于非编码区序列的遗传变异。非编码序列通过参与基因的表达调控而影响表型。如目前在玉米中鉴定到的青花素合成基因b1,株型基因TB1,以及开花期基因ZmCCT9和vgt1,都受到非编码区序列的调控。然而,玉米的非编码区调控元件调控目的基因表达的分子机理目前还不清楚。
真核生物中,顺式调控元件可以与同一染色体上的其他区域(即使线性结构上相隔很远)发生互作,形成DNA环,并调控该位点的基因表达。因此,染色质的互作模式对于解析基因的表达调控非常重要。三维基因组学是一门专门研究染色体空间结构以及染色质互作的学科。常用的研究方法有Hi-C和CHiA-PET等,其中Hi-C是一种非靶向的研究方法,能够研究基因组内所有的互作,但是其分辨率较低;而ChiA-PET能够研究某个特定蛋白所介导的染色质互作,虽然所得到的数据量较Hi-C少,但是分辨率更高,适用于研究某个特定蛋白所介导的染色质互作。

华中农业大学李兴旺教授、严建兵教授和李国亮教授团队合作,首次利用Long-read ChIA-PET技术,解析了玉米活跃表达基因中的顺式调控元件所参与的染色质互作,该结果揭示了玉米染色质互作调控基因的表达进而影响表型变异的潜在机理。华中农业大学博士研究生彭勇和熊丹为论文的共同第一作者。
2、研究内容及结果
➤1.CHiA-PET研究了H3K4me3和RNA聚合酶所介导的基因组远程互作
本研究首先利用RNA聚合酶Ⅱ(RNA Polymerase II)和不同组蛋白修饰的CHiP-seq数据,定义了玉米品种B73的功能性DNA顺式调控元件。并结合RNA-seq数据,研究了这些调控元件在基因表达中的作用。同时利用MNase测序数据和DNA甲基化数据定义了远程顺式调控元件,并鉴定到了远程调控元件中保守的DNA motif。高丰度的H3K4me3修饰和RNAPII结合(RNAPII occupancy)是基因活跃表达的重要特征之一,作者利用H3K4me3抗体(ABclonal提供,A2357)和RNAPII的抗体进行了Long-read ChIA-PET实验,结果鉴定到了H3K4me3所介导的染色质互作49766个,RNAPⅡ介导的染色质互作25002个。综合两个ChIA-PET数据,共检测到28875个基因启动子和基因启动子互作(Promoter proximal region interaction, PPI),9152个远程顺式调控元件和基因启动子的相互作用(promoter-proximal and distal interactions, PDI)。
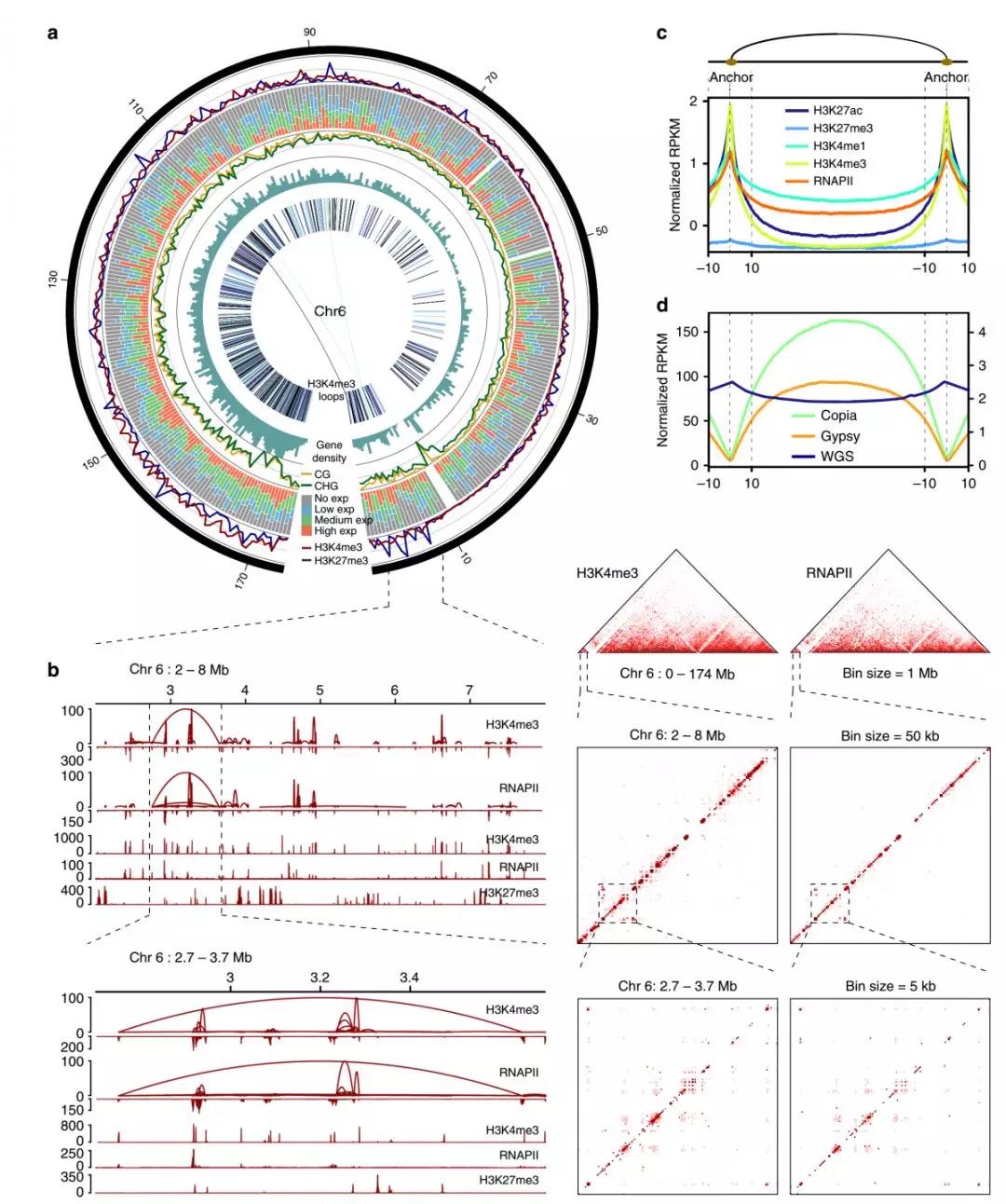
Figure 1. Characterization of RNAPII and H3K4me3 ChIA-PET data in maize seedlings
➤2、eQTL控制eTrait与基因组远程互作有关
将基因的表达水平(eTraits)作为表型进行QTL定位,可以鉴定到控制该eTrait的eQTL,对前期 B73和Mo17重组自交系群体中鉴定到的eQTL和eTrait数据进行了分析,结果显示,eQTL和eTrait发生基因组远程互作的几率显著高于随机区域(Figure 2b,2d),这说明eQTL可能通过基因组远程互作调控基因的表达(eTrait),基于此,作者提出了eQTL调控基因表达的染色质互作模型(Figure 2i)。
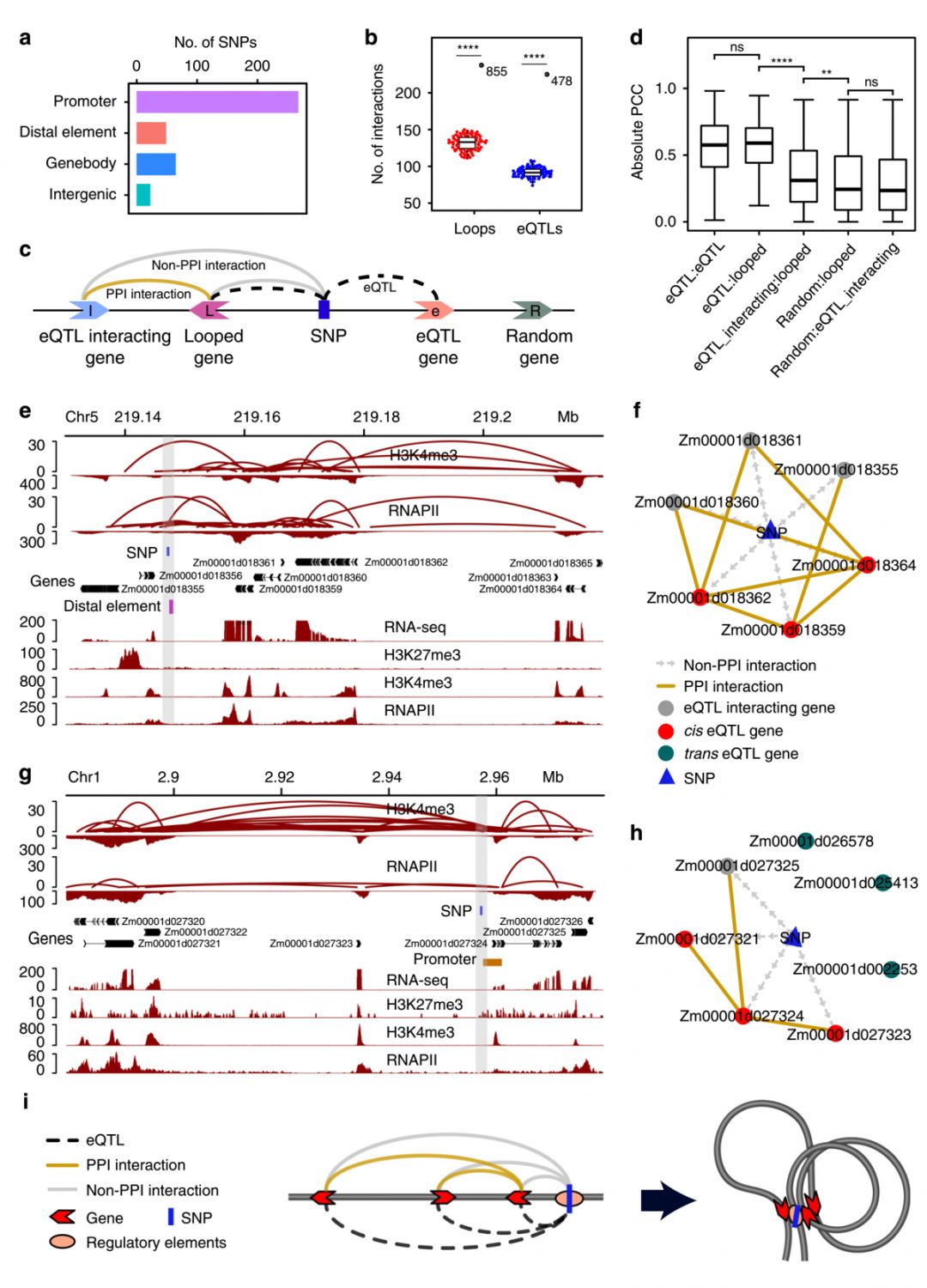
Figure 2. Regulation of eQTLs to their associated genes mediated by chromatin interactions.
➤3、染色质互作影响玉米的表型
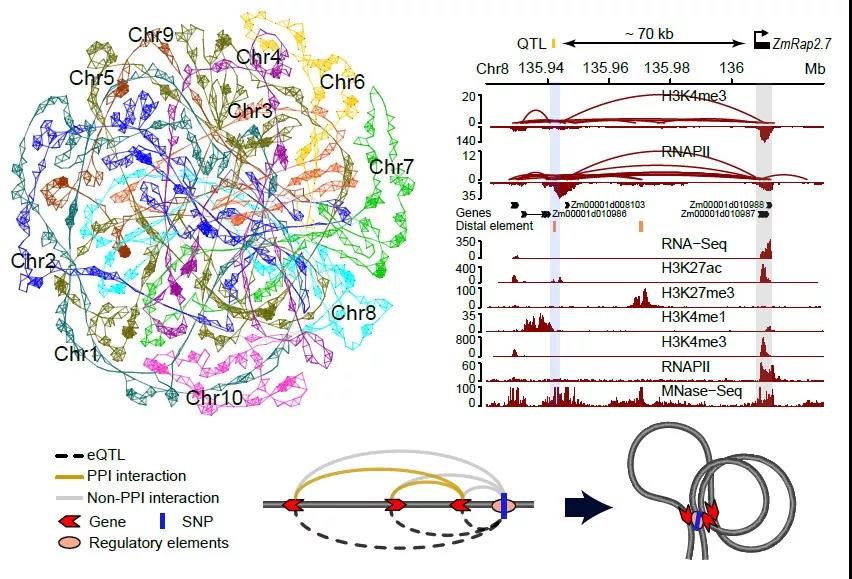
研究人员发现参与染色质互作的DNA顺式调控元件在农艺性状和代谢产物相关QTL中显著富集(Figure 3d)。比如一个开花相关QTL被定位在了开花基因ZmCCT9上游57 Kb处的一个转座子元件处,该转座子能抑制ZmCCT9的表达从而促进开花,而本研究鉴定到了一对分别锚定在ZmCCT9启动子和该转座子远端元件的互作(Figure 3e)。另外,vgt1是另一个被定位在基因间区的开花相关QTL,本研究结果显示,vgt1 附近的一个调控元件能和其上游70 Kb处的ZmRap2.7发生明显互作(Figure 3f)。这些结果表明,玉米基因组中的远端调控元件可能通过染色质互作,影响下游靶基因的表达进而产生表型变异。
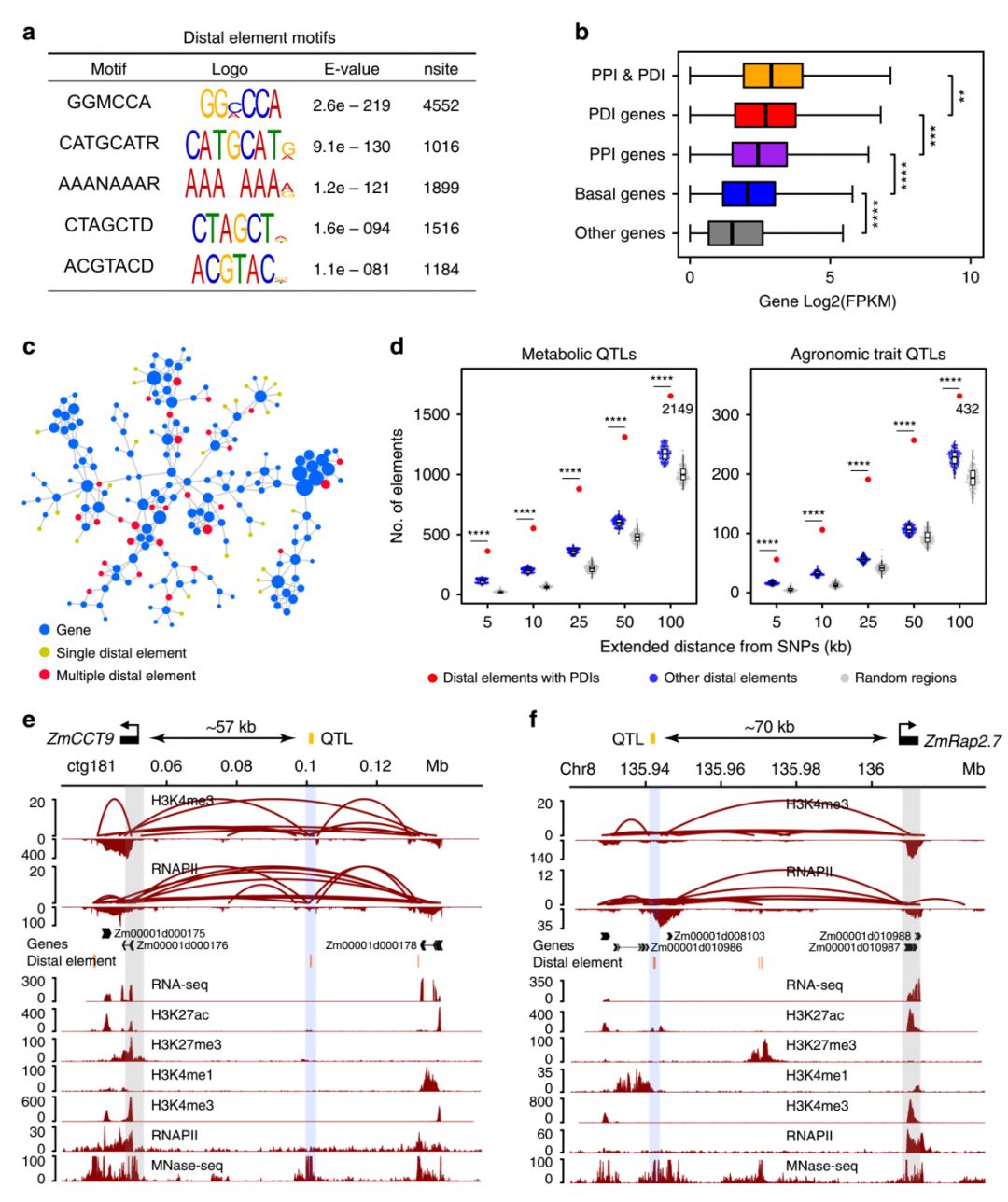
Figure 3. Chromatin loops connect QTLs to the genes causing phenotypic changes.
文章小结
基因的表达调控一直是生命科学领域最重要的研究方向,染色质互作是基因表达调控的重要方式,但是在之前的研究中较少涉及。本研究利用最新的三维基因组的研究方法,绘制了玉米苗期活跃表达基因的顺式元件的三维互作模式,该结果对于玉米基因表达调控研究具有重要作用,进而对玉米复杂数量的解析提供了线索。
3、表观抗体案例展示
A2360 H3K9me3
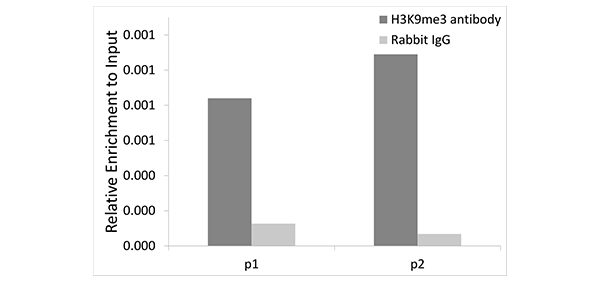
Chromatin immunoprecipitation analysis of extracts of 293T cells, using TriMethyl-Histone H3-K9 antibody (A2360) and rabbit IgG. P1 and P2 were located on EBAG9 gene. The amount of immunoprecipitated DNA was checked by quantitative PCR. Histogram was constructed by the ratios of the immunoprecipitated DNA to the input.
A2366 H3K36me3
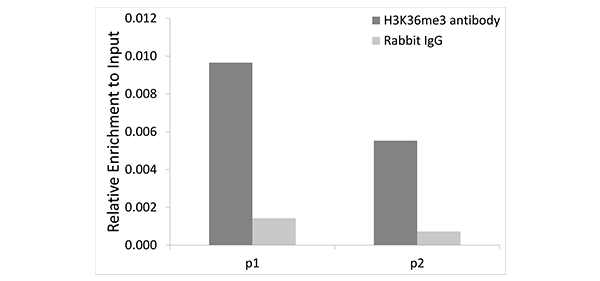
Chromatin immunoprecipitation analysis of extracts of 293T cells, using TriMethyl-Histone H3-K36 antibody (A2366) and rabbit IgG. P1 and P2 were located on GAPDH gene. The amount of immunoprecipitated DNA was checked by quantitative PCR. Histogram was constructed by the ratios of the immunoprecipitated DNA to the input.
A7258 H4K8ac
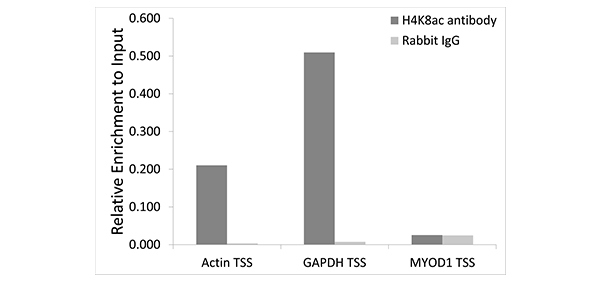
Chromatin immunoprecipitation analysis of extracts of 293 cell line, using H4K8ac antibody (A7258) and rabbit IgG. The amount of immunoprecipitated DNA was checked by quantitative PCR. Histogram was constructed by the ratios of the immunoprecipitated DNA to the input.
参考文献
1. Noonan, J. P. & McCallion, A. S. Genomics of long-range regulatory elements. Annu. Rev. Genomics Hum. Genet. 11, 1–23 (2010).
2. Zhang, W., Garcia, N., Feng, Y., Zhao, H. & Messing, J. Genome-wide histone acetylation correlates with active transcription in maize. Genomics 106, 214–220 (2015).
3. Atlasi, Y. & Stunnenberg, H. G. The interplay of epigenetic marks during stem cell differentiation and development. Nat. Rev. Genet. 18, 643–658 (2017).
4. Oka, R. et al. Genome-wide mapping of transcriptional enhancer candidates using DNA and chromatin features in maize. Genome Biol. 18, 137 (2017).
5. Kieffer-Kwon, K. R. et al. Interactome maps of mouse gene regulatory domains reveal basic principles of transcriptional regulation. Cell 155, 1507–1520 (2013).
6. Mifsud, B. et al. Mapping long-range promoter contacts in human cells with high-resolution capture Hi-C. Nat. Genet. 47, 598–606 (2015).
7. Lieberman-Aiden, E. et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science 326, 289–293 (2009).
8. Li, G. et al. Extensive promoter-centered chromatin interactions provide a topological basis for transcription regulation. Cell 148, 84–98 (2012).
9. Tang, Z. et al. CTCF-mediated human 3D genome architecture reveals chromatin topology for transcription. Cell 163, 1611–1627 (2015).

